Актуальность темы. Цель селекционной работы это, прежде всего, подбор пар для спариваний с высокой племенной ценностью, чтобы в следующем поколении был получен прогнозируемый селекционный успех [5, 6]. Большинство селекционных программ основываются на статистическом анализе больших баз данных по связям между генотипическими и фенотипическими характеристиками животных, на основании которых в дальнейшем делается прогноз развития желательных признаков у носителей определенных генотипов [7, 9, 10]. Однако история исследований в области использования генетических маркеров для таких прогнозов свидетельствует об ограниченных возможностях их прямого внедрения в практические программы разведения [1, 3, 4]. Появление методов геномного картирования высокоплотного распределения мононуклеотидных замен (Single Nucleotide Polymorphisms – SNP), объединенных с новыми статистическими обработками получаемых данных для ранних оценок племенной ценности животных, позволило сформулировать новое направления MAS (markerassistedselection), получившее название «геномная селекция» [2, 8, 13, 17].
Геномная селекция – метод племенной работы, основанный на изучении корреляции набора ДНКмаркеров и племенной ценности животного, оцененной в системе BLUP. Эта методика внедрена в селекционные программы во многих странах мира, она позволяет проводить отбор производителей на ранних этапах их жизни [11].
Геномная селекция является одной из форм MAS-селекции с помощью маркеров, в которой генетические маркеры покрывают геном полностью и все локусы количественных признаков (QTL) оказываются сцеплены хотя бы с одним маркером. Этот подход возможен благодаря высокой распространенности мононуклеотидных замен, обнаруженных при прочтении генома и новых методов эффективного генотипирования по огромному количеству SNP [3, 15].
Известно, что в мире распространена практика внедрения в новые регионы пород сельскохозяйственных животных, показавших себя эффективными в местах происхождения породы (например, голштинизация местных пород) [10, 12, 17]. На акклиматизацию животных и адаптацию пород к новым эколого-климатическим условиям разведения и эксплуатации расходуется большое количество ресурсов. При этом желательным является не только повышение продуктивности, но и совершенствование качества получаемой сельскохозяйственной продукции, снижение количества заболеваний и падежа животных, а также снижение затрат на единицу продукции [17]. Для этого необходим качественно новый подход к ведению племенной работы с целью улучшения генофонда пород [1, 7, 8], использующихся в животноводстве на территории России. Как возможное решение подобных вопросов может выступить применение такой передовой области аграрной науки, как геномная селекция [11, 19, 22].
Результаты моделирования и экспериментов говорят о том, что племенная ценность животного может быть предсказана с высокой точностью с применением только генетических маркеров, однако больше валидации требуется особенно для образцов из популяций, отличных от тех, в которых эффект маркера был оценен [10, 19].
Необходимость выявления различий между генофондами аборигенных и специализированных молочных пород, а также гибридного скота обусловлена необходимостью разработки простых методов контроля генетической консолидированности групп животных, популяционно-генетических преобразований, возникающих в процессе селекционной работы, а также при адаптации животных к разным условиям разведения [12, 14, 21].
Цель работы заключалась в анализе генома различных пород крупного рогатого скота путем выполнения сравнительного анализа спектров продуктов амплификации, основанных на выявленных полилокусных спектрах ISSR-PCR маркеров.
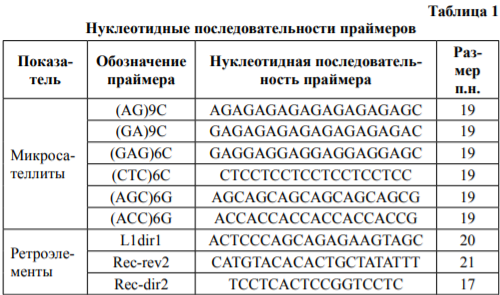
Материал и методы исследований. В настоящей работе выполнен сравнительный анализ спектров, получаемых с использованием в качестве праймеров 6 нуклеотидных последовательностей (AG)9C; (GA)9C; (GAG)6C; (CTC)6C; (AGC)6G; (ACC)6G, и длинного концевого повтора эндогенного ретровируса ERV1-2C-LTR и участков гомологии к ретротранспозону L1-2_BT.
Геномное сканирование было проведено на 124 образцах ДНК животных 9 пород крупного рогатого скота: калмыцкая порода (15 гол., Республика Калмыкия), айрширская порода (25 гол., Московский конный завод № 1), черно-пестрая голштинизированная порода (15 гол., ЗАО «Можайский»; 10 гол., ОАО «Куйбышево», Московская обл.), голштинская порода (10 гол., СПК «Заря», Республика Коми), красная эстонская порода (10 гол., Псковская область), казахская белоголовая порода (9 гол., г. Волгоград), тагильская порода (10 гол.), якутский скот (10 гол., Республика Саха (Якутия)), зебувидный скот (10 гол., «Снегири», Московская обл.). Образцы крови были получены в разные периоды, предшествующие эксперименту.
Геномную ДНК экстрагировали из крови крупного рогатого скота с помощью набора реагентов Magna DNA Prep 200 в соответствии с протоколом производителя. Концентрацию геномной ДНК измеряли с помощью спектрофотометра Eppendorf Bio Photometer Plus; в дальнейшей работе использовали аликвоты ДНК, приведенные к стандартной концентрации 2 ng/mkl.
Амплификацию индивидуальных фрагментов ДНК осуществляли методом ISSR-PCR, с помощью которого амплифицируются участки ДНК, находящиеся в геноме между инвертируемыми повторами используемого праймера. ПЦР проводилась по отработанной ранее методике [8].
В качестве праймеров использовались ди- и тринуклеотидные коровьи мотивы микросателлитов с якорными нуклеотидами: (AG)9C; (GA)9C; (GAG)6C; (CTC)6C; (AGC)6G; (ACC)6G, фрагменты ретротранспозона L1-2_BT (L1dir1 и Rec-rev2) и эндоретровируса ERV1-2C-LTR (Rec-dir2). В табл. 1 приведен нуклеотидный состав праймеров.
На основании присутствия/ отсутствия фрагментов ДНК определенной длины были построены бинарные матрицы. Математическая обработка данных проводилась при помощисервиса Dendro UPGMA [18] и коэффициента подобия по Дайсу. Использовали алгоритм метода невзвешенного попарного среднего UPGMA (unweighted pair-group method using arithmetic averages). Полученные в формате Newick генетические деревья использовались для построения дендрограмм в программе TreeView [20]. Статистическая обработка проводилась в программе GenAlEx 6.5 [21].
Расчет индекса PIC (Poly morphic Information Content) выполнялся по формуле для диаллельных локусов, для которых PIC=2f (1‒f), где f – частота одного из двух аллелей. Поскольку ISSR-PCR маркеры имеют доминантный характер проявления по присутствию продукта амплификации, f рассчитывали по формуле f=√R, где R – частота встречаемости животных среди исследованных, у которых в спектрах продуктов амплификации отсутствовал фрагмент ДНК данной длины. Значение R рассматривалось как доля гомозигот по рецессивному аллелю.
Результаты исследований и их обсуждение. В результате секвенирования генома крупного рогатого скота получены данные о том, что одной из его характеристик, отличающих его от других видов млекопитающих, является повышенная частота встречаемости AGC микросателлита. Причем его высокая частота встречаемости объединяет геномы крупного рогатого скота и овец и отличает их от геномов человека, собаки, крысы. Обнаружено также, что в геноме крупного рогатого скота тандемные повторы AGC с частотой в 39 % ассоциированы с неавтономным транспозоном Bov-A2 (SINE) [16].
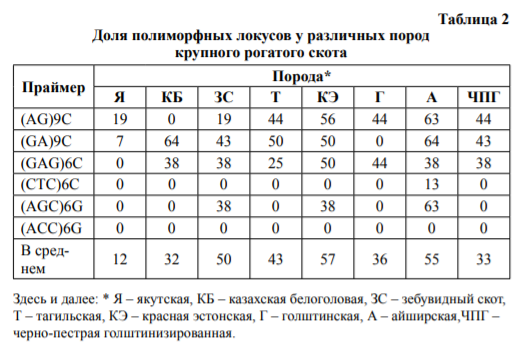
Это позволяет предполагать, что распространенность данного микросателлита ассоциирована с экзогенной ретровирусной инфекцией, специфичной для жвачных и дальнейшего распространения по их геномам в связи с транспозициями мобильных генетических элементов. Можно ожидать, что межпородные отличия по полиморфизму фрагментов ДНК, фланкированных инвертированным повтором этого микросателлита, отражает специфические особенности его вовлеченности в такие транспозиции (табл. 2).
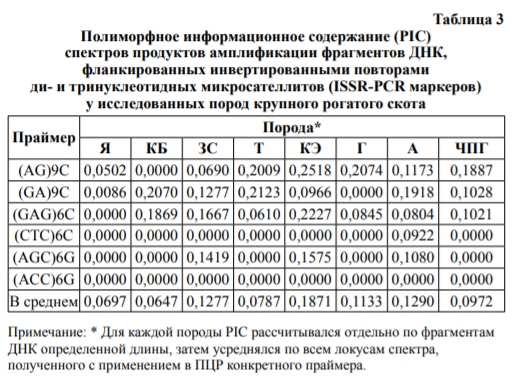
Суммарно оценки полиморфизмов у исследованных групп крупного рогатого скота по полиморфному информационному содержанию (PIC) спектров продуктов амплификации, полученных с использованием в ПЦР всех микросателлитных фрагментов, представлены в табл. 3.
По величинам средних значений PIC по всем спектрам апликонов оказываются близкими представители таких пород, как якутская, казахская белоголовая, тагильская; у представителей остальных пород значения этого показателя заметно выше. Уникальным оказалось полиморфное информационное содержание спектров продуктов амплификации фрагментов ДНК, фланкированных инвертированным повтором (CTC)6C у айширской породы, составляющее 0,0922 против 0,0000 у других групп животных.
Спектры всех праймеров можно условно подразделить на легкие фрагменты продуктов амплификации длиной от 200 до 500 пар оснований (п.о.), средние – длиной от 600 до 1400 п.о., тяжелые – от 700 до 2500 п.о. В спектрах праймера (AG)9C выделялся зебувидный скот, только у его представителей присутствовали в спектрах ампликонов фрагменты ДНК длиной от 200 до 300 пар нуклеотидов; у всех других животных фрагменты таких длин отсутствовали. По спектрам средних и тяжелых зон выраженных отличий между молочными и другими породами не наблюдалось.
В спектрах продуктов амплификации, полученных с праймером (GA)9C, существенных отличий между представителями двух групп пород (1-я специализированные молочные, 2-я – аборигенные породы и зебувидный скот) не обнаружено. В спектрах продуктов амплификации с праймером (GAG)6C только у животных специализированных молочных пород обнаруживалась зона длиной в 750 п.о., которая отсутствовала у представителей всех других пород.
В спектрах продуктов амплификации, полученных с праймером (CTC)6C, не выявлено существенных отличий между представителями двух групп пород (за исключением айрширской).
В спектрах продуктов амплификации праймера (AGC)6G также не выявлено существенных отличий между представителями двух групп пород, как и в спектрах, полученных с праймером (CTC)6C. Исключение представляют две легкие зоны длиной в 200 и 250 п.о., присутствующие только у зебувидного скота.
В спектрах праймера (ACC)6G именно у пород специализированного молочного скота наблюдается присутствие легких фрагментов длиной в 200 и 300 п.о., в отличие от зебувидного скота, тагильской и казахской белоголовой пород. Причем те же зоны выявлены у якутского скота, что представляет особый интерес в связи с тем, что небольшая якутская корова по количеству общего удоя относительно живой массы производит достаточно высокое количество молока по сравнению с многими другими аборигенными отечественными породами.
Полученные данные свидетельствуют о том, что группы молочных пород отличаются от аборигенных не столько присутствием в спектрах продуктов амплификации определенных фрагментов ДНК, не выявляемых у других пород, сколько их сочетанием. По преобладанию легких зон в спектрах, полученных с такими праймерами, как (AG)9C и (AGC)6G, зебувидный скот отличался от всех остальных животных.
Специализированные молочные породы отличались от остальных пород по спектру праймера (GAG)6C присутствием фрагмента ДНК длиной в 750 п.о., и от трех аборигенных пород (тагильская, казахская белоголовая, якутский скот) – присутствием легких зон длиной в 200 и 300 п.о. в спектрах праймера (ACC)6G.
Представленные результаты исследований свидетельствуют о том, что каждая порода крупного рогатого скота имеет свои специфические особенности полиморфизма спектра ампликонов, выявляемых с применением в ПЦР разных микросателлитов в качестве праймеров (ISSR-PCR маркеры). Это приводит к необходимости предварительных исследований результативности их подбора для конкретных групп животных при разработках тестсистем для геномного сканирования, эффективность которого непосредственно зависит от количества полиморфных локусов и уровня их полиморфизма. На основании выполненных исследований можно ожидать, что для голштинского и голштинизированного скота наиболее полиморфные спектры ампликонов можно получить при использовании в ПЦР в качестве праймеров динуклеотидные микросателлиты, как (AG)9C и (GA)9C, для других пород – еще и (AGC)6G, а для айширской – последовательность (CTC)6C.
Уникальность сочетаний продуктов амплификации ДНК, полученных с использованием разных праймеров, обнаруживается и при построении дендрограммы (с помощью программы TFPGA) на основании величин генетических расстояний М. Нея, рассчитанных по частотам встречаемости фрагментов ДНК разных длин у исследованных пород крупного рогатого скота (рис.).
Дендрограммы, построенные по генетическим расстояниям, рассчитанным суммарно по всем выявленным микросателлитным ISSR-PCR маркерам, подтверждают общепринятую дифференциацию между специализированными молочными породами и другими породами. Данная дендрограмма указывает на полиморфизм на генетическом уровне у животных различного направления продуктивности.
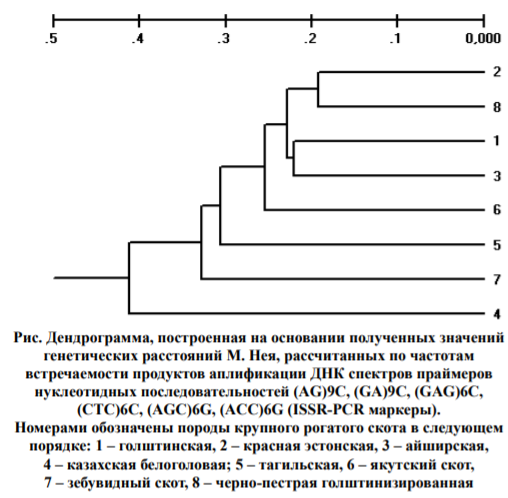
На рисунке представлен общий кластер, имеющий 2 подкластера. Первый кластер образуется подкластером, состоящим из групп красной эстонской породы, чернопестрой голштинизированной породы и второго подкластера из голштинской породы и айширской породы. В общем, все четыре группы этого подкластера представляют специализированный скот молочного направления продуктивности. Остальные группы животных являются представителями разного направления продуктивности и менее специализированы, но традиционно с более высоким адаптивным потенциалом (4 – казахская белоголовая, 5 – тагильская порода, 6 – якутский скот, 7 – зебувидный скот).
Выявленная межпородная дифференциация по совокупности генотипов, оцененных на основании спектров продуктов амплификации, полученных в ПЦР с использованием в качестве праймеров различных микросателлитных локусов, отражает особенности распределения инвертированных повторов в исследуемых геномных ДНК в диапазоне длин в 2000 п.о. Дендрограмма, построенная на основании оценок полиморфизма ISSR-PCR, свидетельствует о тесной связи между выявленным полиморфизмом по исследованным фрагментам ДНК и дифференциацией генофондов животных по направлению и уровню специализации.
Выводы
1. Генетические взаимоотношения, выявленные на основании анализа распределения и полиморфизма длин участков ДНК, фланкированных инвертированными повторами ENV1_BT, существенно различаются у специализированных молочных пород и казахской белоголовой породы, однако при этом характеризуются низкой внутрипородной изменчивостью.
2. Относительно низкая внутрипородная изменчивость позволяет рекомендовать полилокусное генотипирование с использованием участков ENV1_BT в качестве праймера для оценки генетической дифференциации пород, достаточно давно разошедшихся от общего корня.
3. Распределение инвертированных повторов участков гомологии к L1_BT имеет относительно повышенную внутрипородную изменчивость. Это может быть обусловлено повышенной транспозиционной активностью L1_BT и использоваться при изучении индивидуальной внутрипородной изменчивости в целях геномной селекции.
4. Полученные данные позволяют создавать и рекомендовать породоспецифичные тест-системы, основанные на выявленных полилокусных спектрах ISSR-PCR маркеров, облегчающие отбор и подбор животных для получения от них потомства с желательным фенотипом, а также направленно контролировать консолидацию групп животных с учетом особенностей их происхождения и породной принадлежности.
КОРОТКО О ВАЖНОМ
ПРОИЗВОДИТЕЛИ МОЛОКА ПРОСЯТ ИЗМЕНИТЬ ПРАВИЛА ПЕРЕХОДА НА ЭВС
Производители молока и продукции из него отмечают, что работа системы электронной ветеринарной сертификации (ЭВС) не отлажена, и ее необходимо протестировать на сырье, прежде чем внедрять электронную сертификацию на готовую продукцию. Опрошенные участники рынка приветствуют переход с бумажных на электронные сертификаты на сырье, но отмечают ряд трудностей, в т. ч. с доступом в Интернет в отдаленных селах.
При этом ЭВС для готовой продукции, по их мнению, в принципе избыточна, и в итоге затраты, которые понесут заводы на увеличение штата сотрудников, их обучение и закупку техники могут привести к росту себестоимости и цен.
С 1 января 2018 г. в РФ вводится обязательная ветеринарная сертификация широкого ряда подконтрольных продуктов животного происхождения, в т. ч. молочной продукции. Она будет осуществляться в государственной информационной системе «Меркурий».
Любое перемещение партии готовой продукции станет возможным только при условии оформления ветсертификата. После ввода ЭВС любое перемещение партии продукции станет возможным только при условии оформления электронного документа. Одним из плюсов возможности отследить продукцию от фермы до прилавка называют борьбу с фальсификатом молока. По данным Роспотребнадзора, его доля в РФ колеблется в районе 6–8 %.
Между тем, как говорит исполнительный директор Российского союза предприятий молочной отрасли Л. Маницкая, на рынке этот процент может быть существенно выше. «Мы надеемся, что программа ЭВС и прослеживаемости полностью решит эту проблему. Там будет все: откуда пришло сырье, что мы применили, чтобы произвести конкретную продукцию, – сколько молока, сколько других составляющих», – сказала она.
Источник: http://tass.ru


