Актуальность темы. Казахской белоголовой породе принадлежит большая роль в становлении и развитии отрасли специализированного мясного скотоводства в Казахстане. Казахская белоголовая порода выведена путем скрещивания казахской и калмыцкой пород с использованием генофонда породы герефорд, что позволяет успешно разводить ее во всех регионах республики, сохраняет к ней стабильный интерес с точки зрения наследственного потенциала продуктивности [2]. Поэтому исследования, направленные на ускорение темпов селекции казахской белоголовой породы, имеют большое значение для Республики Казахстан.
Одним из наиболее перспективных современных методов, применяемых для идентификации локусов, контролирующих количественные признаки продуктивности у крупного рогатого скота, является метод полногеномного анализа ассоциаций (Genome-Wide Association, GWA), который позволяет установить связь между аллельной вариацией в некотором регионе генома и исследуемым признаком [1, 8].
Для регистрации singlenucleotide polymorphism (SNP) в ДНК в настоящее время доступны чипы низкой (около 10 тыс. SNP), средней (более 50 тыс. SNP) и высокой (более 800 тыс. SNP) для оценки наиболее важных в хозяйственном отношении видов животных [10]. Однако такие исследования являются дорогостоящими для внедрения в практику рутинной оценки продуктивности поголовья крупного рогатого скота. Поэтому актуальность сохраняет маркирование признаков продуктивности по небольшому количеству SNP, с известными фенотипическими эффектами, которые можно объединить в небольшие диагности ческие ДНК-панели, доступные по стоимости.
В данной статье представлены результаты поиска породоспецифичных для казахской белоголовой породы генетических маркеров повышенной мясной продуктивности на основе данных ДНК-типирования животных с помощью чипа GeneSeek GGP Bovine 150K (Illumina Inc., США).
Цель исследований – установить породоспецифичные SNP-маркеры мясной продуктивности крупного рогатого скота казахской белоголовой породы.
Материал и методика исследований. Для проведения молекулярно-генетических исследований использовались волосяные луковицы, отобранные от 100 гол. крупного рогатого скота казахской белоголовой породы. Геномную ДНК экстрагировали и генотипировали в Neogen Agrigenomics, США, согласно протоколу производителя GeneSeek GGP Bovine 150K (Illumina Inc., США).
Контроль качества и фильтрацию данных генотипирования для каждого SNP и каждого образца выполняли с использованием программного пакета PLINK 1.9 (http:// zzz.bwh.harvard.edu/plink/) [4, 10].
Поиск уникальных областей генома проводили с помощью построения рядов гомозиготности (ROH) по PLINK Cattle QTL database [4].
Для выявления геномных адресов SNP-гаплотипов, связанных с признаками мясной продуктивности, оценивали частоту отдельных SNP у каждой особи, полученный результат анализировали с помощью манхэттенского графика [3, 6, 7], на котором пики с повышенной частотой свечения соответствуют наиболее часто встречающимся в данном участке SNP у данной породы. По геномным координатам выявленных породоспецифичных участков генома был проведен поиск QTL, заявленных для этих пород по базе данных Cattle QTL Database [9]. Затем в этих участках через базу данных Ensembl.org были получены списки генов, локализованных в данных QTL и некоторые их характеристики. Для характеристики генов-кандидатов использовались данные открытой международной генетической базы http://www.pantherdb. org [11].
Поиск SNP, локализованных в генах-кандидатах, пригодных для включения в небольшие ДНКпанели, подходящие для массового скрининга поголовья и локализованные в областях потенциальных генов-кандидатов, осуществлен с помощью международной базы данных http://www.ensembl. org [12].
Результаты исследований и их обсуждение. Поиск уникальных областей генома с помощью построения рядов гомозиготности (ROH) [5] и манхэттенский график выявили геномные адреса породоспецифичных SNP-гаплотипов на следующих хромосомах: 2 – участки 67226923:72583890, 73165500: 74246725 и 74322634:74444101, 5 – участок 17125373:19048571, 6 – участок 64466274:88186013, 13 – участки 64228423:64660314 и 13:65140902:65236809, 14 – участок 81915831:82174926, 26 – участок 48407133:50450382 п.н.
С помощью открытой электронной базы http://www.ensembl.org [12] по полученным геномным QTL, ассоциированных с мясной продуктивностью, выявлено 24 генакандидата, локализованных в породоспецифичных областях генома крупного рогатого скота казахской белоголовой породы (табл. 1).
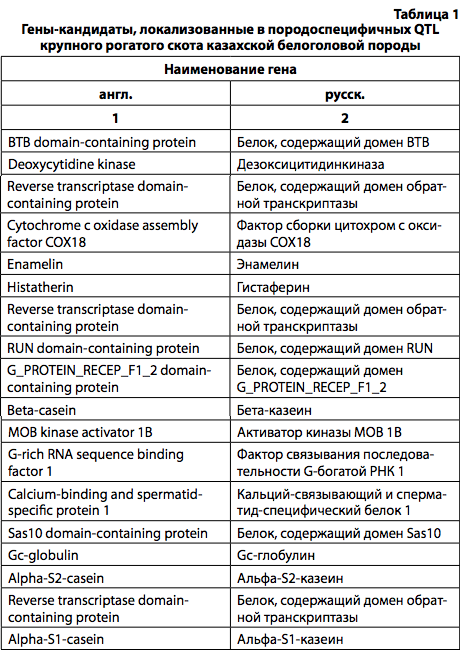
В результате поиска по базе http://www.ensembl.org [12] для этих генов было выделено 12 SNP, которые находились либо в регуляторных областях, либо в экзонах и приводили к аминокислотной замене в структуре белка.
На следующем этапе была проведена идентификация отобранных SNP с положением на чипе и оценена частота встречаемости этих SNP в группах животных с разной продуктивностью. Из 12 SNP, отобранных по базе данных http:// www.ensembl.org [12], на чипе присутствовало только 10. Для них были подсчитаны частоты встречаемости и проведен сравнительный анализ у животных класса элита и элита-рекорд с показателем частот этих полиморфизмов с группой животных I–II классов и ниже (табл. 2).
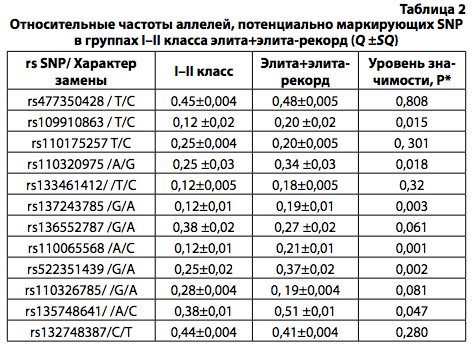
Примечание. * различие между группами значимо при Р < 0,05.
Как видно из данных табл. 2 в качестве SNP-маркеров повышенной мясной продуктивности у крупного рогатого скота казахской белоголовой породы можно рассматривать:
1) аллель С (rs109910863) в гене гистоферина (histatherin (HSTN));
2) аллель С (rs110065568) в гене миостатина (myostatin (MSTN);
3) аллель G (rs110320975) в гене гистоферина (histatherin (HSTN));
4) аллель С (rs135748641) в гене нуклеолярного протеина, взаимодействующего с доменом FHA фактора MKI67 (nucleolar protein interacting with the FHA domain of MKI67 (NIFK);
5) аллель G (rs136552787) в гене инсулиноподобного фактора, связывающего протеин 6 (insulin like growth factor binding protein 6 (IGFBP6–202);
6) аллель А (rs137243785) в гене гистоферина (histatherin (HSTN201));
7) аллель А (rs522351439) в гене миостатина (myostatin (MSTN)).
Заключение. В результате проведенных исследований мы пришли к следующим выводам:
1. Проведены исследования с помощью биочипа GeneSeek GGP Bovine 150K со средней плотностью покрытия 150000 SNP (Illumina Inc., США), прогенотипировали 999 животных казахской белоголовой породы. Всего выявлено 119841 SNP полиморфизмов, подходящих для полногеномного ассоциативного поиска генов роста и развития животных.
2. Определены уникальные SNP-гаплотипы, соответствующие потенциальным QTL у крупного рогатого скота казахской белоголовой породы, обнаружены на хромосомах: 2 – участки 67226923:72583890, 73165500: 74246725 и 74322634:74444101, 5 – участок 17125373:19048571, 6 – участок 64466274:88186013, 13 – участки 64228423:64660314 и 13:65140902:65236809, 14 – участок 81915831:82174926, 26 – участок 48407133:50450382 п.н.
3. Выявлено 7 SNP-маркеров повышенной мясной продуктивности у крупного рогатого скота казахской белоголовой породы: аллель С (rs109910863) в гене гистоферина (histatherin (HSTN)), аллель С (rs110065568) в гене миостатина (myostatin (MSTN), аллель G (rs110320975) в гене гистоферина (histatherin (HSTN)), аллель С (rs135748641) в гене нуклеолярного протеина, взаимодействующего с доменом FHA фактора MKI67 (nucleolar protein interacting with the FHA domain of MKI67 (NIFK), аллель G (rs136552787) в гене инсулиноподобного фактора, связывающего протеин 6 (insulin like growth factor binding protein 6 (IGFBP6–202)), аллель А (rs137243785) в гене гистоферина (histatherin (HSTN-201)), аллель А (rs522351439) в гене миостатина (myostatin (MSTN)).
Работа выполнена в рамках грантового финансирования фундаментальных и прикладных научных исследований молодых ученых по научным и (или) научно-техническим проектам Министерства образования и науки Республики Казахстан AP08052960 «Породоспецифичное QTL-маркирование мясной продуктивности крупного рогатого скота аулиекольской и казахской белоголовой пород на основе полногеномного SNP-чипирования» (№ госрегистрации 0120RK00043).


